【深度学习】详解Resampling和softmax模型集成
【深度学习】详解Resampling和softmax模型集成

文章目录
1 图像重采样
1.1 次级采样(sub-sampling)
1.2 高斯金字塔(Gaussian pyramids)
1.3 上采样(upsampling)
2 医学图像预处理之重采样
3 医疗图像重采样代码分析
4 softmax集成
5 在图像分割中用于多通道conv2d输出的Sigmoid或Softmax1 图像重采样
图像重采样包含两种情形,一种是下采样(downsampling),把图像变小;另一种是上采样(upsampling),把图像变大。
1.1 次级采样(sub-sampling)
每隔一个,扔掉行和列,创建一个更小的图像。

下采样(downsampling)
根据Nyquist采样定律,采样频率大于等于2倍的图像的最大频率。

对于高清图片,如果直接采样,采样频率很高。
如果先对图像进行模糊化处理(高斯滤波),就可以降低采样频率了,
最后进行次级采样(sub-sampling),就可以得到小一倍的图片了。
总结:下采样=高斯滤波+次级采样

1.2 高斯金字塔(Gaussian pyramids)
高斯模糊是什么
模糊算法,不论是使用哪种算法,目的都是为了让图片看起来不如原来那么清晰。
清晰的图片,像素间的过渡会较为干脆利落,简而言之,就是像素之间的差距比较大。
而模糊的本质,其实就是使用某种算法把图像像素和像素之间的差距缩小,让中间点和周围点变得差不多;即,让中间点取一个范围内的平均值。
模糊到了极致,比如用于计算模糊的取值区域为整张图片,就会得到一张全图所有像素颜色都差不多的图片:
高斯金字塔实际上是图像的多尺度表示法。模仿人眼在近处看到的图像细致,对应金字塔底层;在远处看到图像较为模糊,但可以看到整个轮廓,对应金字塔顶层。
1.3 上采样(upsampling)
插值(interpolation):离散信号之间插入新的值。
但实际情况,函数F[x]是未知的。
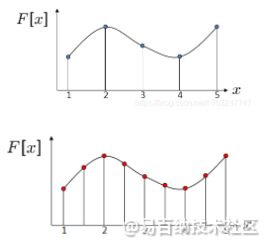
图像的插值也可以通过构造函数与图像的卷积运算实现。插入的新值即为新的像素色度值。
对于相同的离散信号,使用不同的构造函数,最后得到的结果也不一样。如下图所示,
图像的实际效果

当对滤波要求较高时,重采样是常见的图像运算之一。重采样就是改变图像大小。
高像素调整为低像素显示的两种方法:
看作是删除像素。在留下的像素之间去掉一个或者两个像素。可以实现压缩图像的目的,但是图像质量变低。
对图像重采样。首先根据输入样本重构一个连续函数,然后对函数进行采样。为了避免走样,在每一步均要选择合适的滤波器。
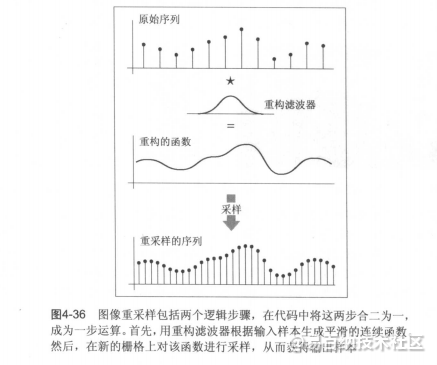
例如下图示例,原始图像为12×9像素,而新图像是8×6像素。在每一维,输出像素数是输入像素数的 2/3,因此图像上的像素间隔是原始图像的 3/2。
2 医学图像预处理之重采样
对于不同的数据类型重采样的方法和目的都不相同。例如在遥感中,重采样是从高分辨率遥感影像中提取出低分辨率影像的过程;在数据挖掘中,重采样是指为了解决训练数据类别不均衡,通过在训练期间通过增加小样本的数量或者减少大样本的数量保持样本类别均衡的算法;在医疗图像中,重采样是指将医疗图像中大小不同的体素归一化到相同的大小。体素是体积元素(Volume Pixel)的简称,一张3D医疗图像可以看成是由若干个体素构成的,体素是一张3D医疗图像在空间上的最小单元,本文主要包含两个部分:第一、通过python和SimpleITK实现医疗图像重采样,并对结果进行分析;第二是讲解一下医疗图像重采样所代表的现实意义。本文所使用的数据集为公开的胰腺分割数据集,其包含82个病人的胰腺数据集。
医疗图像重采样
对于一张大小为128128的彩色图像,其在计算机中可以表示为1281283的矩阵,其中每一个像素点的取值范围为0-255,不同的数值代表不同的亮度。但是对于医疗图像其是由若干个slice组成的,假设每一个slice的大小为512512的单通道的图像,其中每一个像素点表示的是一个体素的取值,其范围可以-1000~2000之间。接下来通过以胰腺分割数据集中PANCREAS_0015.nii.gz为例,对医疗图像中体素这个概念进行讲解。Spacing(0.78125, 0.78125, 1.0)表示的是原始图像体素的大小,也可以将Spacing想象成大小为(0.78125, 0.78125, 1.0)的长方体。而原始图像的Size为 (512, 512, 247),表示的是原始在X轴,Y轴,Z轴中体素的个数。原始图像的size对应的Spacing既可以得到真实3D图像大小(5120.78125,5120.78125,2471 ),在图像重采样只是修改体素的大小,而真实3D图像大小是保持不变的,因此假设我们将Spacing修改成(1.0, 1.0, 2.0)的时候,则修改之后其对应的size应该为((5120.78125)/ 1.0,(5120.78125)/ 1.0,(247*1 ))即(400, 400, 124)。结果如下所示。
3 医疗图像重采样代码分析
对于医疗图像重采样,可以分成三个步骤:首先使用SimpleITK读取数据,获得原始图像的对应的Spacing以及Size,得到图像原始的大小;然后图像原始的大小除以新Spacing得到新Size;最后将新新Spacing得到新Size赋值到读取的数据即可。
# 对医疗图像进行重采样,仅仅需要将out_spacing替换成自己想要的输出即可
def resample_image(itk_image, out_spacing=[1.0, 1.0, 2.0]):
original_spacing = itk_image.GetSpacing()
original_size = itk_image.GetSize()
# 根据输出out_spacing设置新的size
out_size = [
int(np.round(original_size[0] * original_spacing[0] / out_spacing[0])),
int(np.round(original_size[1] * original_spacing[1] / out_spacing[1])),
int(np.round(original_size[2] * original_spacing[2] / out_spacing[2]))
]
resample = sitk.ResampleImageFilter()
resample.SetOutputSpacing(out_spacing)
resample.SetSize(out_size)
resample.SetOutputDirection(itk_image.GetDirection())
resample.SetOutputOrigin(itk_image.GetOrigin())
resample.SetTransform(sitk.Transform())
resample.SetDefaultPixelValue(itk_image.GetPixelIDValue())
resample.SetInterpolator(sitk.sitkBSpline)
return resample.Execute(itk_image)
gz_path = 'PANCREAS_0015.nii.gz'
print('测试文件名为:', gz_path)
# 使用sitk读取对应的数据
Original_img = sitk.ReadImage(gz_path)
print('原始图像的Spacing:', Original_img.GetSpacing())
print('原始图像的Size:', Original_img.GetSize())
# 对数据进行重采样
Resample_img = resample_image(Original_img)
print('经过resample之后图像的Spacing是:', Resample_img.GetSpacing())
print('经过resample之后图像的Size是:', Resample_img.GetSize())
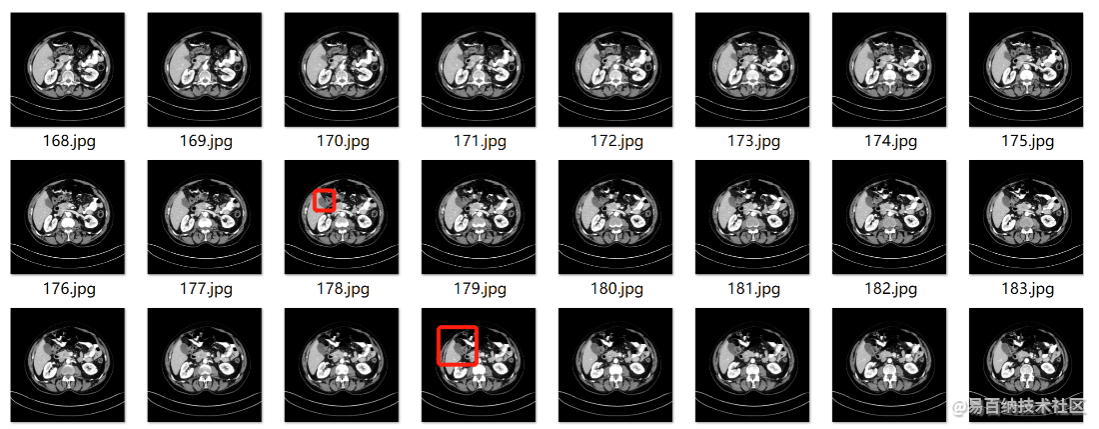
重采样之前
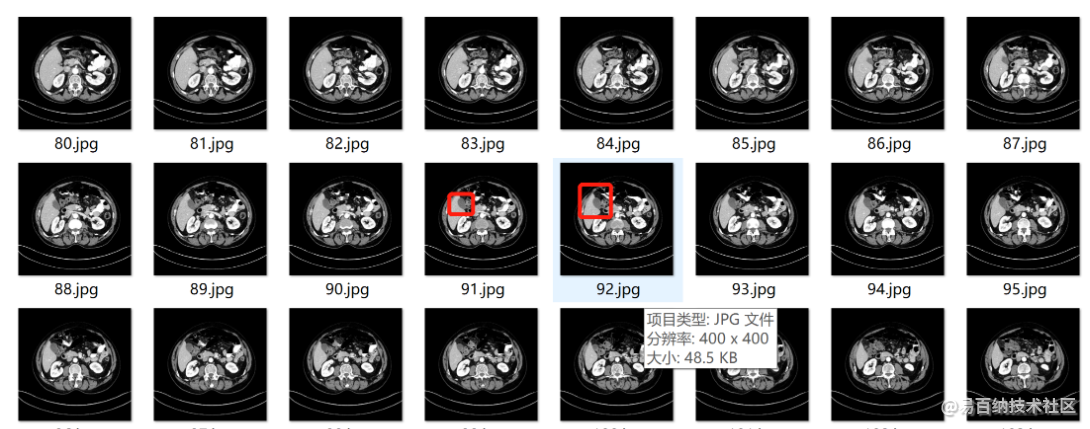
重采样之后
针对第一个问题:在我看到的资料中一般是在构建一个3D医疗图像模型时,使用图像图重采样技术,对此知乎上的一个解释可以仅供参考,CNN中Conv操作被提出来的其中一个重要motivation就是图像中有相似的块能用共享的卷积来提取特征,因此对所有图像重采样能减少不同图像之间的不一致性,便于卷积操作提取共同的特征
针对第二个问题:我个人感觉,重采样中Spacing的设置就是一个超参。对于这个超参数,我的想法是新设置的Spacing应该尽可能使得不同图像之间的体素差异性尽可能的小,差异越小效果越好。在2D图像中,我更喜欢保持X轴,Y轴尽可能的不变,而把Z轴变小,这样不会影响图像的分辨率,而且能够增加训练数据集。
4 softmax集成
Unweighted Averaging:
CNNs标准的集成方法是未加权的平均。每个模型的softmax概率进行平均,来得到最后的预测结果。
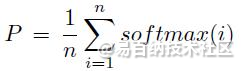
5 在图像分割中用于多通道conv2d输出的Sigmoid或Softmax
我正在训练我的U架构进行像素分类。 我有9个班,所有班级都是互斥的。 每个标签用0到9的不同像素值表示,包括背景,并且标签之间没有重叠。
因此,我将其视为多类分类。
我在U-net的最后一层是
out=layers.conv2D(num_classes, Activation='softmax')(previous_layer)
model.compile(optimizer=Adam(lr), loss='categorical_crossentropy', metrics=[mean_iou])我检查了Softmax激活如何将给出每个类和输出和的概率绑定到一个。
但是,如果我不考虑背景类并且有8个类,为什么不能使用Sigmoid? 。 例子:
out=layers.conv2D(num_classes, Activation='sigmoid')(previous_layer)
model.compile(optimizer=Adam(lr), loss='binary_crossentropy', metrics=[mean_iou])如果这也行得通,那么为什么对所有多类问题都使用Softmax?
- 分享
- 举报
 暂无数据
暂无数据-
浏览量:14754次2021-06-15 10:27:34
-
浏览量:5576次2021-06-21 11:50:25
-
浏览量:5362次2021-08-05 09:21:07
-
浏览量:5528次2021-08-05 09:20:49
-
浏览量:4857次2023-09-04 14:32:32
-
浏览量:7817次2021-06-15 10:28:29
-
浏览量:5371次2021-04-21 17:06:33
-
浏览量:1044次2023-09-18 15:02:26
-
浏览量:5674次2021-06-07 09:28:15
-
浏览量:4838次2021-04-09 16:28:04
-
浏览量:6583次2021-08-03 11:36:37
-
浏览量:6411次2021-08-03 11:36:18
-
浏览量:6244次2021-07-15 10:44:33
-
浏览量:1936次2023-02-13 10:31:50
-
浏览量:19868次2021-07-15 10:45:21
-
浏览量:7037次2021-06-07 09:26:53
-
浏览量:7209次2021-04-19 14:56:57
-
浏览量:10768次2021-06-15 10:30:15
-
浏览量:6624次2021-06-11 12:41:01
-
广告/SPAM
-
恶意灌水
-
违规内容
-
文不对题
-
重复发帖
这把我C





 微信支付
微信支付举报类型
- 内容涉黄/赌/毒
- 内容侵权/抄袭
- 政治相关
- 涉嫌广告
- 侮辱谩骂
- 其他
详细说明


 微信扫码分享
微信扫码分享 QQ好友
QQ好友





